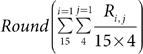세포형태 분석의 한계
중추신경계(CNS)의 신경세포 및 미세아교세포는 다양한 병리 상태에 따라 미세한 형태 변화를 겪습니다. 특히, 신경염증(neuroinflammation)은 이러한 형태 변화의 중요한 원인이며, 이를 정량적으로 분석하는 것은 뇌 질환 연구와 치료제 개발에 있어 매우 중요한 작업입니다. 하지만, 이러한 분석 대부분은 현미경 이미지의 수작업 분석에 의존하며, 이로 인해 정량성과 재현성이 크게 제한되었습니다.
또한, 현재 사용되는 세포 기반 분석법은 염증 정도나 약물 반응을 정량화하는 데 있어 높은 해상도와 민감도가 요구되며, 전문가에 의한 판단은 주관성과 처리량의 한계를 동반합니다. 더욱이, 실험 간 조건 차이에 의한 배치 효과(batch effect)가 결과에 큰 영향을 미칠 수 있으며, 이는 대규모 스크리닝 실험의 신뢰성을 저하시킵니다. (심한 경우에는 그날의 오존 농도에 따라 다른 결과가 있을 수 있다는 논문도 있습니다...)
딥러닝 기반의 분석
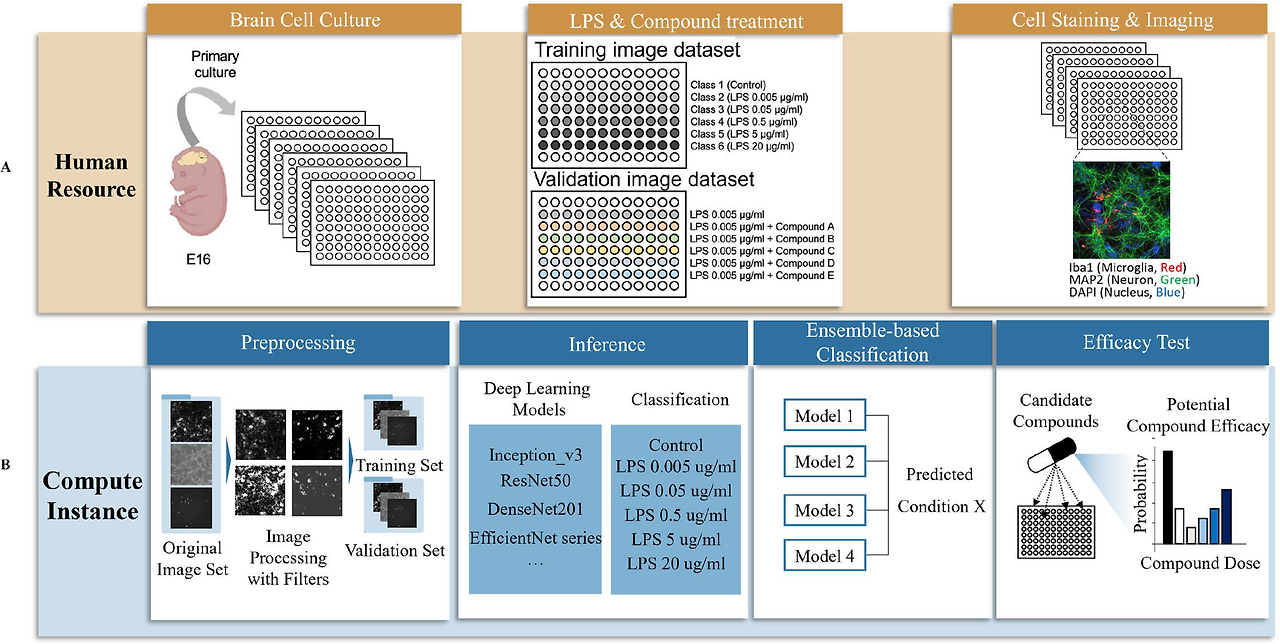
이 연구는 위와 같은 문제를 해결하기 위해 딥러닝을 통한 세포 이미지 분석 파이프라인을 구축하였습니다. 뇌에서 유래한 1차 배양 세포를 다양한 농도의 LPS로 처리하여 염증 상태를 인위적으로 연출하고, 세포를 면역염색하여 형광 현미경 이미지를 획득한 후, 이를 딥러닝 모델에 학습시켰습니다.
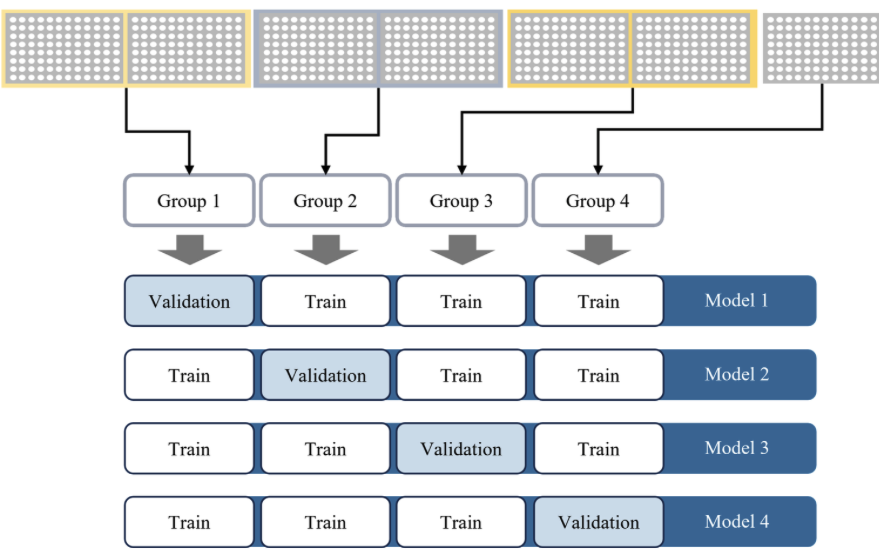
특히 실험 간 배치 효과를 줄이기 위해, 일반적인 cross-validation이 아닌 `plate 단위의 leave-plates-out 교차 검증기법`을 도입하였고, 더 나아가 `앙상블 모델(ensemble model)`을 사용하여 여러 plate 기반 모델의 예측값을 평균화함으로 예측의 안정성과 일반화 성능을 높였습니다. 이러한 접근은 모델 성능 향상과 실제 약물 스크리닝에서 발생할 수 있는 실험 간 편차를 줄이는 데 중요한 역할을 하였습니다.
모델링
결국 이미지 자료다 보니 CNNs 아니면 ViTs 패밀리 중 어떤 구조를 선택하느냐입니다. 본 연구에서는 대표적으로 CNNs: EfficientNet, ResNet, DenseNet, Inception-v3 와 ViTs: Swin-V2, MaxViT, ConvNeXt에 대한 실험을 진행하였습니다.
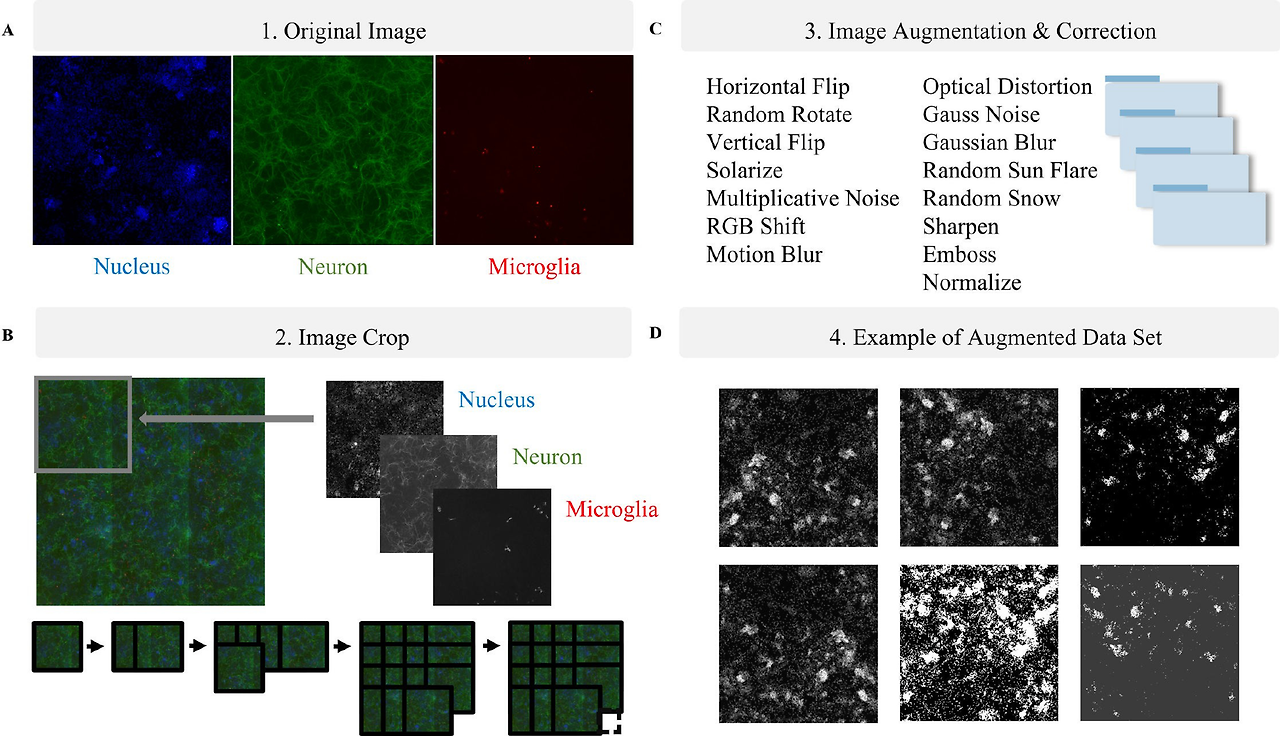
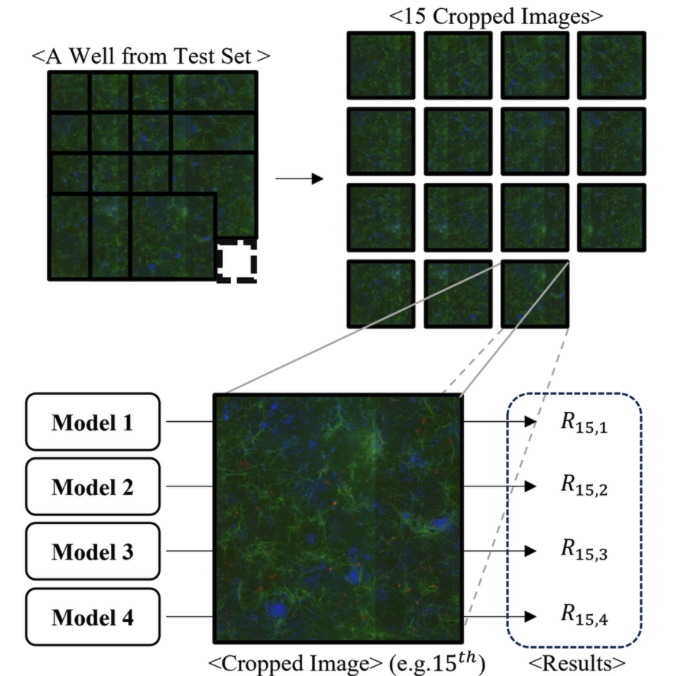
모든 모델은 동일한 데이터셋(총 3,150장의 3채널 이미지, plate 7개)을 기반으로 학습되었으며, 성능 평가는 주로 F1-score를 사용하였습니다. 실험실의 조건으로 하나의 well은 9조각으로 나누어 촬영되며 테두리의 36 wells는 촬영시 positional confound factor가 우려되어 padding으로 두었습니다. 9조각을 이어 붙인 각 well은 15개의 이미지 패치로 구성되며, 여러 증강기법을 적용시킵니다. 그 이후 모델은 각각의 이미지를 독립적으로 분류한 후 이를 종합하여 well 단위의 예측을 수행합니다.
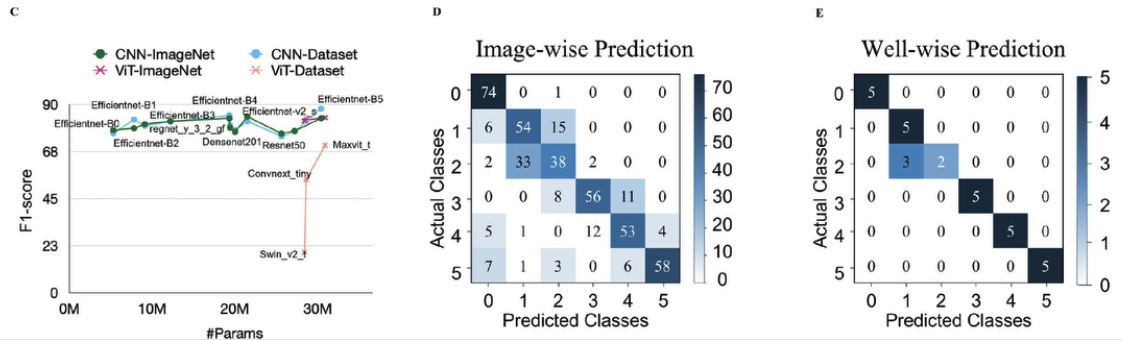
그 결과, EfficientNet-B5가 가장 높은 F1-score (88%)를 기록하였고, ViT 계열은 ImageNet에서는 우수한 성능을 보였으나 이 실험에서는 CNN보다 낮은 성능을 보였습니다. 이는 세포 이미지처럼 local feature가 중요한 데이터에서는 CNN이 여전히 우위에 있다는 점을 시사하는 것 같습니다만... 조직 이미지를 썻다면 또 모를까, 사실 transformer를 쓰기에는 이미지 수가 아주 많이 더 필요하지 않았나 싶습니다.
이 모델에 더해 배치효과를 완화하는 방식을 적용하였습니다. 개인적으로는 이 논문의 Kick이지 않을까 싶습니다. 먼저 `Leave-plates-out-cross-validation` 을 통해 4개의 모델을 만듭니다.
그 후 하나의 cropped image에 대해 각 모델의 예측 결과를 `앙상블`하여 결과를 냅니다.
이렇게 나온 각각의 결과에 대하여 아래의 식을 통해 하나의 well에 대한 예측을 합니다.
결과
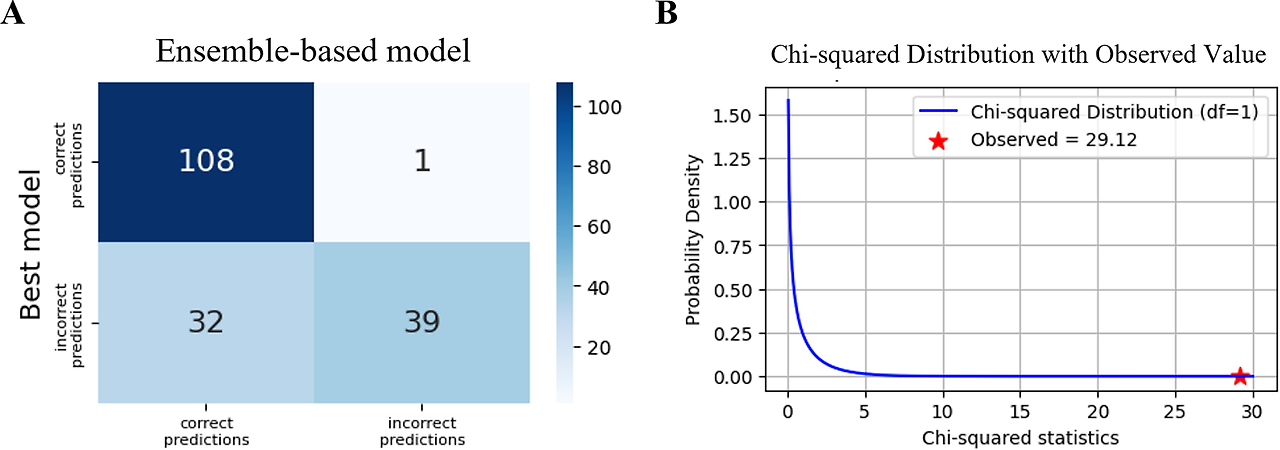
모델 성능 평가 결과, 앙상블 모델은 단일 모델 대비 더욱 우수한 예측력을 보였습니다. 특히, Control vs LPS(0.005μg/ml) 처리된 이미지 180장을 대상으로 평가했을 때, 앙상블 모델의 F1-score는 77%, 단일 최고 성능 모델은 69%를 기록하였습니다.
추가로, 이 차이를 검정하기 위해 McNemar’s Test를 수행하였으며, χ² = 27.27, p < 0.001이라는 매우 유의미한 통계 결과를 통해 앙상블 방식과 단일 모델의 결과는 유의미한 차이가 있다는 결론에 도달합니다. 아래 테이블 A를 확인해보면 실제로 단일 모델에 비해 앙상블 모델이 잘못 비교하는 경우가 현저히 적습니다(32>1). 앙상블 방식이 우수하다고 충분히 해석 가능한 부분입니다.
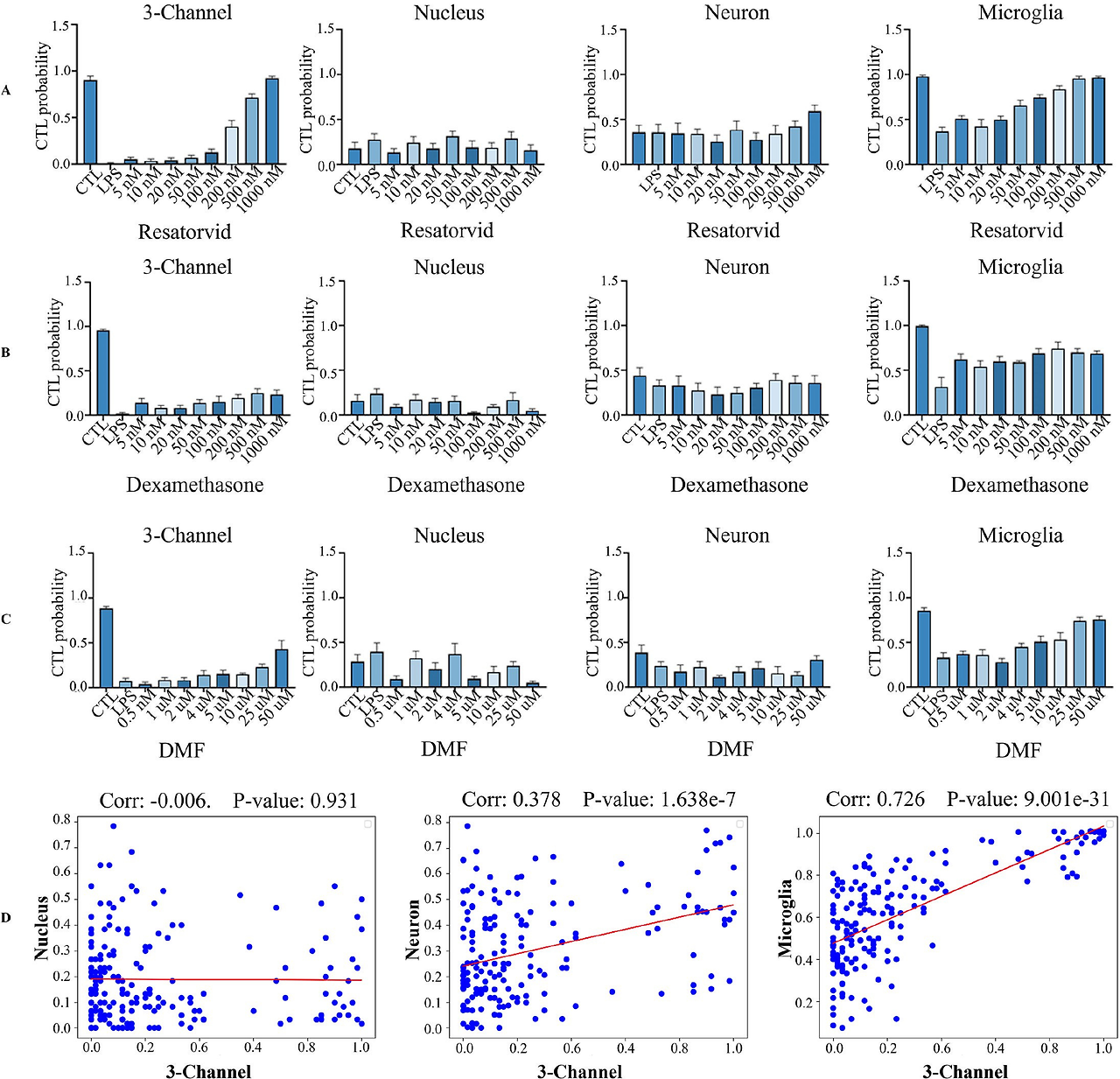
모델의 실용성은 실제 약물 스크리닝 실험을 통해 확인되었습니다. Resatorvid, Dexamethasone, DMF 등의 항염증 약물을 다양한 농도로 처리한 후, 세포가 '정상 상태'로 회복되는 정도를 예측하였으며, 약물 농도 증가에 따라 Control로 분류되는 이미지 비율이 증가하는 dose-response 관계가 확인되었습니다. 특히, Iba-1 (미세아교세포 마커) 채널의 예측 결과는 3채널 전체 예측과 높은 상관관계($r > 0.7$)를 보이며, 이는 미세아교세포가 신경염증 평가에 매우 중요한 생물학적 지표임을 시사한다고 볼 수 있습니다.
기여
비록 이 프로젝트에 늦게 합류하긴 했지만, 이미지 자료에 대한 뛰어난 지식을 지닌 동료들이 많아 배우고자 하는 마음으로 참여하였습니다. 그래서인지 이 논문에서는 제가 주요하게 기여한 부분이 적고, 잔잔바리로 참여했습니다. 최종 디테일한 수정, 서브미션 가이드라인 맞추기, McNemar's test 적용 및 해석 추가, method & ensemble 부분의 writing, GitHub 작성 정도가 되겠군요! 이 프로젝트에서 더 해야할 부분이 있다면, 데이터가 쌓이는대로 ViTs를 적용해볼것과 torch를 JAX로 바꿔 시간을 빠르게 해본다는것 정도가 있을듯 합니다.
논문 정보
https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0320204
Unveiling CNS cell morphology with deep learning: A gateway to anti-inflammatory compound screening
Deciphering the complex relationships between cellular morphology and phenotypic manifestations is crucial for understanding cell behavior, particularly in the context of neuropathological states. Despite its importance, the application of advanced image a
journals.plos.org
https://github.com/DRNOAHBIOTECH/NeuroRG
GitHub - DRNOAHBIOTECH/NeuroRG: Scripts for the research paper "Unveiling CNS Cell Morphology with Deep Learning: A Gateway to A
Scripts for the research paper "Unveiling CNS Cell Morphology with Deep Learning: A Gateway to Anti-Inflammatory Compound Screening" - DRNOAHBIOTECH/NeuroRG
github.com
'논문참여' 카테고리의 다른 글
| [co-first] SF-Rx: A Multi-output Deep Neural Network-Based Framework Predicting Drug-Drug Interaction (0) | 2025.05.30 |
|---|